[ad_1]
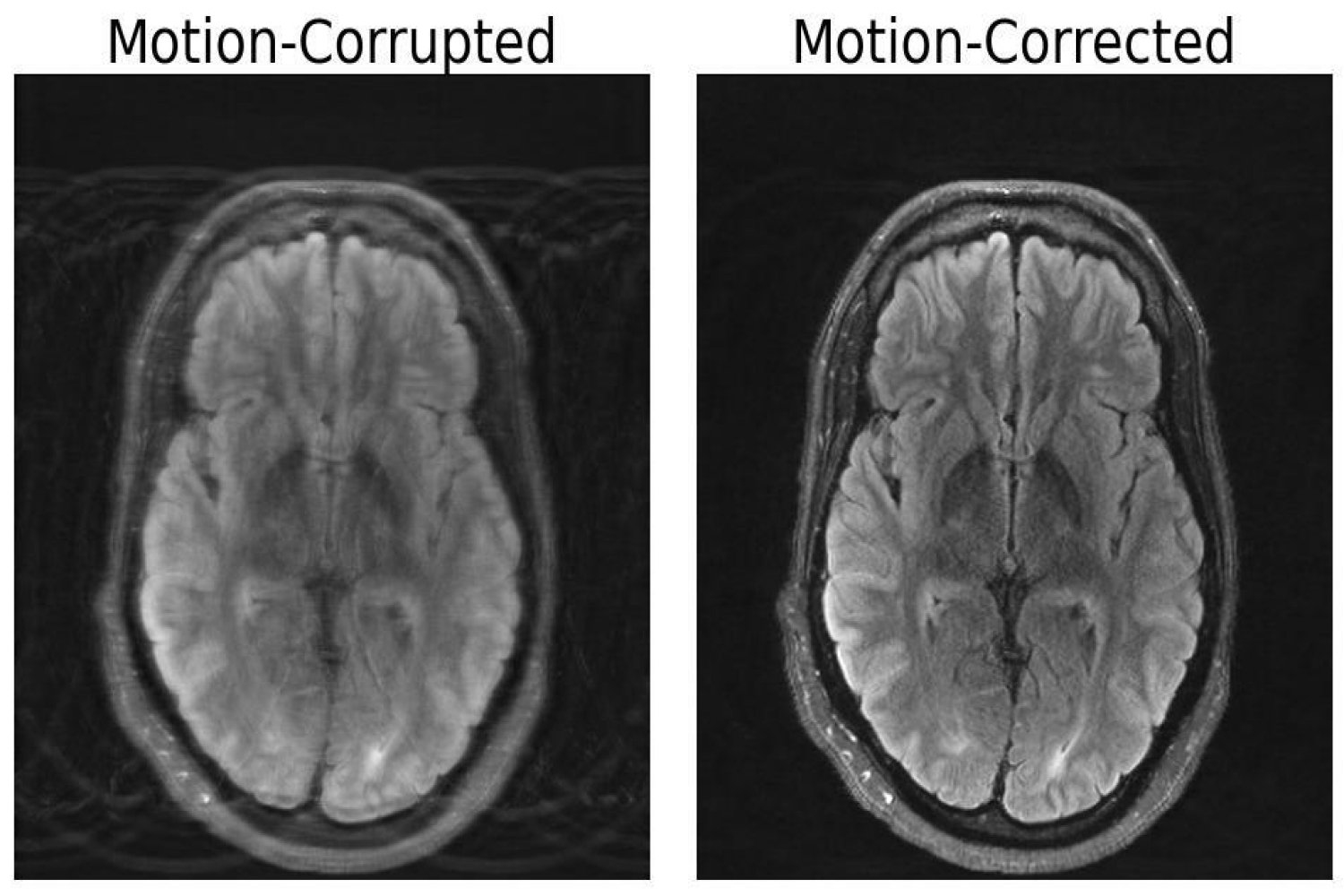
Rispetto ad altre modalità di imaging come i raggi X o le scansioni TC, le scansioni MRI forniscono un contrasto dei tessuti molli di alta qualità. Sfortunatamente, la risonanza magnetica è altamente sensibile al movimento, con anche il più piccolo dei movimenti che provoca artefatti dell’immagine. Questi artefatti mettono i pazienti a rischio di diagnosi errate o di trattamenti inappropriati quando i dettagli critici vengono nascosti al medico. Ma i ricercatori del MIT potrebbero aver sviluppato un modello di apprendimento profondo in grado di correggere il movimento nella risonanza magnetica cerebrale.
“Il movimento è un problema comune nella risonanza magnetica”, spiega Nalini Singh, uno studente di dottorato affiliato alla Abdul Latif Jameel Clinic for Machine Learning in Health (Jameel Clinic) nel programma Harvard-MIT in Health Sciences and Technology (HST) e autore principale di la carta. “È una modalità di imaging piuttosto lenta.”
Le sessioni di risonanza magnetica possono richiedere da pochi minuti a un’ora, a seconda del tipo di immagini richieste. Anche durante le scansioni più brevi, piccoli movimenti possono avere effetti drammatici sull’immagine risultante. A differenza dell’imaging della fotocamera, in cui il movimento si manifesta tipicamente come una sfocatura localizzata, il movimento nella risonanza magnetica spesso si traduce in artefatti che possono corrompere l’intera immagine. I pazienti possono essere anestetizzati o invitati a limitare la respirazione profonda per ridurre al minimo il movimento. Tuttavia, queste misure spesso non possono essere prese in popolazioni particolarmente suscettibili al movimento, inclusi bambini e pazienti con disturbi psichiatrici.
Il documento, intitolato “Data Consistent Deep Rigid MRI Motion Correction”, è stato recentemente premiato come migliore presentazione orale alla conferenza Medical Imaging with Deep Learning (MIDL) a Nashville, nel Tennessee. Il metodo costruisce computazionalmente un’immagine priva di movimento da dati danneggiati dal movimento senza modificare nulla della procedura di scansione. “Il nostro obiettivo era combinare la modellazione basata sulla fisica e il deep learning per ottenere il meglio da entrambi i mondi”, afferma Singh.
L’importanza di questo approccio combinato risiede nell’assicurare la coerenza tra l’output dell’immagine e le misurazioni effettive di ciò che viene rappresentato, altrimenti il modello crea “allucinazioni” – immagini che sembrano realistiche, ma sono fisicamente e spazialmente imprecise, potenzialmente peggiorando i risultati quando arriva alle diagnosi.
L’ottenimento di una risonanza magnetica priva di artefatti da movimento, in particolare da pazienti con disturbi neurologici che causano movimenti involontari, come il morbo di Alzheimer o il morbo di Parkinson, trarrebbe benefici non solo per i risultati dei pazienti. Uno studio del Dipartimento di radiologia dell’Università di Washington ha stimato che il movimento colpisce il 15% delle risonanze magnetiche cerebrali. Il movimento in tutti i tipi di risonanza magnetica che porta a ripetute scansioni o sessioni di imaging per ottenere immagini di qualità sufficiente per la diagnosi si traduce in circa $ 115.000 di spese ospedaliere per scanner su base annua.
Secondo Singh, il lavoro futuro potrebbe esplorare tipi più sofisticati di movimento della testa e movimento in altre parti del corpo. Ad esempio, la risonanza magnetica fetale soffre di movimenti rapidi e imprevedibili che non possono essere modellati solo da semplici traslazioni e rotazioni.
“Questa linea di lavoro di Singh e compagnia è il prossimo passo nella correzione del movimento MRI. Non solo è un eccellente lavoro di ricerca, ma credo che questi metodi saranno utilizzati in tutti i tipi di casi clinici: bambini e anziani che non riescono a stare fermi davanti allo scanner, patologie che inducono il movimento, studi sui tessuti in movimento, persino pazienti sani si muoverà nel magnete”, afferma Daniel Moyer, assistente professore alla Vanderbilt University. “In futuro, penso che probabilmente sarà una pratica standard elaborare immagini con qualcosa che discenda direttamente da questa ricerca”.
I coautori di questo articolo includono Nalini Singh, Neel Dey, Malte Hoffmann, Bruce Fischl, Elfar Adalsteinsson, Robert Frost, Adrian Dalca e Polina Golland. Questa ricerca è stata supportata in parte da GE Healthcare e dall’hardware computazionale fornito dal Massachusetts Life Sciences Center. Il team di ricerca ringrazia Steve Cauley per le utili discussioni. Ulteriore supporto è stato fornito da NIH NIBIB, NIA, NIMH, NINDS, Blueprint for Neuroscience Research, parte del progetto multiistituzionale Human Connectome, BRAIN Initiative Cell Census Network e Google PhD Fellowship.
[ad_2]
Source link

